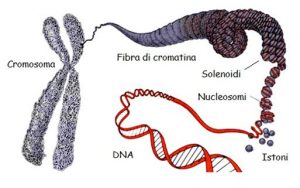
Il DNA ( Acido Desossiribonucleico) è la molecola con cui è stato scritto il genoma, il progetto molecolare che, attraverso un sofisticato alfabeto fatto di monomeri detti nucleotidi, contiene le informazioni biosintetiche relative al funzionamento degli organismi viventi. Al fondamentale ruolo che ricopre il DNA consegue l’interesse di tutti i biologi, che nel corso del XX secolo sono riusciti nel’impresa di decodificarne il complesso linguaggio, attraverso studi che sono collimati nel Progetto ENCODE, l’enciclopedia internazionale del DNA.
Progetto ENCODE: tutto ma proprio tutto sul DNA
All’alba del III millennio nasce il Progetto ENCODE, ideato dall’ente di ricerca statunitense National Human Genome Research Istitute (NHGRI), con lo scopo di determinare e catalogare tutti gli elementi del genoma di organismi modello, in maniera estesa ed enciclopedica.
Lo stesso Ente di ricerca, nato nel 1989, pubblica il 15 febbraio del 2001 la mappa del genoma umano, ricostruita grazie ad un minuzioso lavoro di sequenziamento. Coronato questo obiettivo, il NHGRI ha voluto alzare l’asticella, impegnandosi in un progetto di ricerca che ha per scopo l’attribuzione di una funzione precisa e discreta ad ogni singolo pezzo di DNA.
Svelati i segreti nascosti tra i solchi della doppia elica che racchiude il programma genetico di homo sapiens, i ricercatori hanno cozzato contro il problema di cercare gli elementi che regolano il DNA stesso e la sua traduzione in proteine; capire il ruolo degli RNA codificanti e di quelli non codificanti; determinare tutti gli enzimi e i fattori di trascrizione coinvolti nel processo di sintesi, e snocciolarne i gradi di interazione, nonché i siti di legame. Non per ultimo: restano da chiarire tanti interrogativi circa la cromatina, forma strutturale nella quale il DNA, associato a proteine, si trova la maggior parte del tempo.
Per sperare di riuscire a vedere un giorno l’ambizioso traguardo l’Istituto ha avuto bisogno di espandersi, chiamando a raccolta tantissimi scienziati da ogni parte del mondo, fondando nel 2003 un consorzio internazionale, che vede impegnati ben 150 stati: il progetto ENCODE, appunto.
Prendendo come organismi modello l’uomo e il topo, ENCODE vuole creare una banca dati nella quale saranno riportati tutti gli esperimenti svolti per individuare gli elementi di regolazione e i tipi di splicing avvenuti in trascrizione, arrivando a decodificare il trascrittoma.
Progetto ENCODE: tecnologia al servizio della conoscenza
Per studiare i complessi meccanismi che regolano il fenomeno della trascrizione cellulare – numerosissime reazioni che avvengono nel microscopico nucleo cellulare, in un spazio temporale ridottissimo – c’è bisogno di un massiccio impiego di sofisticata tecnologia da laboratorio.
La trascrizione è alla base del differenziamento cellulare. Ogni cellula infatti possiede al suo interno il genoma completo, ma ne esprime solo una piccola parte: quella necessaria a svolgere il peculiare compito per il quale si sono specializzate. Attraverso la trascrizione vengono selezionate le porzioni di genoma da tradurre successivamente in proteoma.
Il progetto ENCODE ha utilizzato nel corso dei suoi esperimenti 147 tipi cellulari differenti, in modo tale da avere un’idea ampia e statisticamente rilevante circa i geni espressi, in termini di qualità e quantità, e per quanto concerne la loro localizzazione nei tessuti.
Per capire quali sequenze di DNA vengano legate dai fattori trascrizionali si può utilizzare la ChIP (Chromatine Immunoprecipitation Assay), trattando le cellule con formaldeide, per forzare le proteine a legarsi alle basi azotate, ed estraendo il DNA in seguito alla reazione, per poi frammentarlo con ultrasuoni. Per il riconoscimento si usano gli anticorpi, molecole in grado di effettuare legami specifici con le proteine. L’ultimo step è l’immunoprecipitazione, altamente selettiva grazie all’uso degli anticorpi.
https://www.youtube.com/watch?v=4oFdS9EN9Pk
Anche l’EMSA può tornare utile a riguardo. Questa tecnica prevede l’uso di un gel, sul quale far migrare il DNA, marcato con isotopi radioattivi, ed elettricità. Il DNA legato a proteine si muove sul gel in maniera differente rispetto a quello nudo, permettendo di discernerli.
L’EMSA è possibile però solo se a monte si conoscono le proteine coinvolte. In caso contrario si usa il footprinting, sostanzialmente simile, ma che prevede una differente marcatura del DNA: un’estremità della sequenza è nuda; all’altra sono aggiunte le proteine. Ad entrambe sono legati fosfati radioattivi. A questo punto di taglia il DNA con la nucleasi I, che taglia i filamenti solo in zone libere da interazioni con altre proteine. Solo a questo punto di effettua l’elettro-migrazione su gel.
Progetto ENCODE: le scoperte più rilevanti
Se al NHGIR si attribuisce la realizzazione del colossale studio Human Genome Project, al progetto ENCODE, consorzio ancora relativamente giovane e quasi all’inizio del percorso che si è prefissato, vanno i meriti di aver fatto luce su moltissimi aspetti circa la struttura della cromatina.
Gli studi fatti sugli RNA di regolazione hanno dimostrato che questi si trovano sugli introni, porzioni di geni che non codificano per proteine (a differenza degli esoni), e che durante il processo di maturazione RNA messagero – filamento ribonucleotidico incaricato di portare fuori dal nucleo le istruzioni genetiche – vengono “scartate” attraverso lo splicing.
Queste scoperte sono concettualmente assai rilevanti, perchè scardinano alla base il vecchio modello di DNA, colpevole di portare ancora avanti desuete idee come quella delle “sequenze spazzatura“. Se è vero che solo una piccola parte del genoma è trascritto con lo scopo di produrre proteine, non vuol dire che il resto si inutile. Una discreta porzione è strutturale, ma buonissima parte lavora nel lavoro di regolazione.
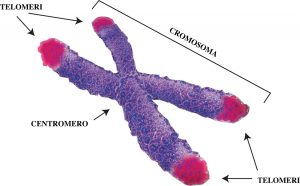 Gli elementi strutturali, come i centromeri e i telomeri, rivestono un ruolo importantissimo nel proteggere i cromosomi e favorirne la duplicazione. Il progetto ENCODE ha scoperto che le sequenze genetiche devolute alla strutturazione desossiribonucleatidica sono ripetute, dalla scarsa complessità genetica.
Gli elementi strutturali, come i centromeri e i telomeri, rivestono un ruolo importantissimo nel proteggere i cromosomi e favorirne la duplicazione. Il progetto ENCODE ha scoperto che le sequenze genetiche devolute alla strutturazione desossiribonucleatidica sono ripetute, dalla scarsa complessità genetica.
Il progetto ENCODE ha inoltre scoperto 400 000 sequenze enancher, tratti di DNA che regolano l’accessibilità della cromatina ai complessi di trascrizione; e 70 000 sequenze promotore, parti di filamento dove comincia lo stesso processo.
Ha inoltre lavorato sullo splicing, scoprendo che un gene eucariotica ha in media 6 modi diversi di organizzare una diversa sequenza attraverso la cucitura tra esone/esone.
E siamo solo all’inizio!
Lorenzo Di Meglio
Bibliografia
D.P. Snustad, M.J. Simmons – Principi di genetica – EdiSES
F. Amaldi et al. – Biologia molecolare – Zanichelli
Link utili